Mejora en la determinación del HH2
Consultorio genómico publicado en la revista Frisona Española 266 (mar-abr 2025)
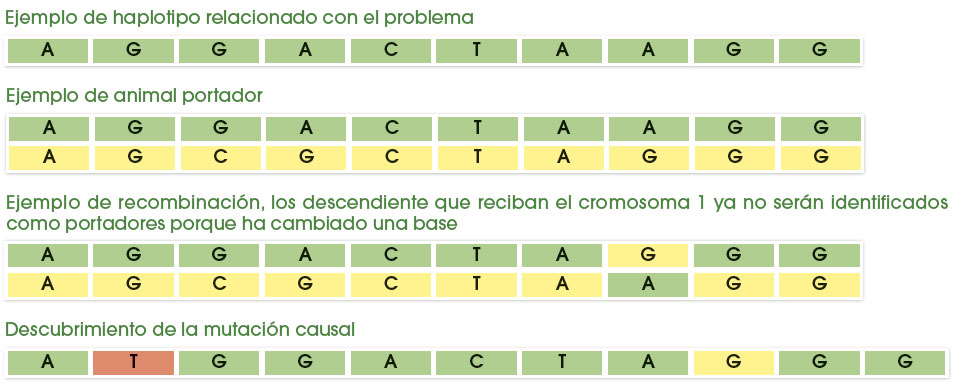
En los últimos meses algunos ganaderos habrán comprobado cómo aparecían animales portadores de HH2 (haplotipo que afecta a la fertilidad) hijos de padres que estaban evaluados como libres. Esta circunstancia anómala se debe a la mejora en el proceso de identificación de este test genético, que ha pasado de ser un Haplotipo a tener incorporado un marcador específico ligado a la mutación concreta.
Al inicio de la genómica, se identificaron en la raza varias regiones para las cuales existían secuencias de bases que se repetían en un porcentaje importante de la población, pero nunca aparecían animales homocigotos (VanRaden et. al 2011). De esta manera se comenzó el estudio del origen de esta anomalía y así surgieron los HH1, HH2... En el número 199 de Frisona Española* ya comentamos estos casos.
Haplotipos que afectan a la fertilidad en la población holstein
A partir de esta información, distintos grupos de investigación fueron descubriendo el origen de la ausencia de homocigotos, que era debida a la inviabilidad de los individuos que recibían esa misma secuencia por parte de su padre y de su madre.
Más adelante se identificó el problema que ocasionaba dicha inviabilidad –por ejemplo, que no se sintetizase alguna proteína esencial para la continuidad del desarrollo embrionario–, y a continuación se fue detectando dónde estaba la mutación que ocasionaba este problema en base a la secuenciación del genoma completo de algunos animales (Adams et al 2012; Fritz et al 2013).
En pocos años las mutaciones se fueron incorporando a los chips de genotipado, pero en el caso del HH2, identificar la posición exacta de la mutación ha llevado más tiempo que el resto (Ortega et al. 2022). Durante este tiempo, se han producido algunos cambios (recombinaciones) en la región identificada inicialmente. Estos cambios suponían que el “haplotipo” fuese ligeramente diferente al usado como referencia y, por tanto, los animales no se identificaban como portadores (del haplotipo original); sin embargo, ahora con la mutación sí se verifica que algunos de los descendientes de los primeros animales identificados (Mark Anthony en este caso) sí tenían el marcador asociado al problema.
Hoy en día todos los test genéticos se basan en marcadores concretos ligados a la mutación, salvo el HH0, para el que se usa la mejor aproximación disponible en la actualidad. Para algunas características como el color, la kappa caseína o el gen polled intervienen más de una posición por lo que también es necesario usar combinaciones de SNPs.
Las personas interesadas en la bibliografía de este artículo pueden solicitarla escribiendo a conafe@conafe.com, referencia Bibliografía HH2
* José Antonio Jiménez, dpto. Técnico de CONAFE. Haplotipos que afectan a la fertilidad en la población Holstein. Frisona Española nº 199, Enero/Febrero 2014, 30-31.
Si quieres leer el artículo en formato PDF, puedes descargarlo desde este enlace y también desde "Documentos".
Artículo publicado por el Departamento Técnico de CONAFE en el número 266 de la revista Frisona Española, correspondiente a los meses de marzo y abril de 2025.
Enlaces
-
YoutubeSuscríbete al canal de YouTube de CONAFE - Frisona Española
-
FacebookSíguenos en la página en Facebook de CONAFE - Frisona Española
-
TwitterSíguenos en el Twitter de CONAFE - Frisona Española
-
InstagramSíguenos en el Instagram de CONAFE - Frisona Española